新闻中心
NEWS
生信篇 | 测序数据比对后处理工具【Mosdepth】
生信专题
测序数据比对后处理
”
Mosdepth是一款快速计算全基因组测序覆盖度的工具,专用于评估 BAM 或 CRAM 文件中基因组的每个碱基位置或基因组区域的深度,常用来评估单条染色体的覆盖深度。
1

优势
1、每个碱基深度的计算速度是传统samtools depth的约2倍。
2、可计算给定窗口大小的平均每个窗口深度,非常方便后续CNV calling。
3、可计算给定区域的BND文件的每个区域的平均值。
4、在设定相应的阈值后,可以展示基因组和每个染色体的碱基分布比例。
5、可以合并相邻碱基的量化输出。
6、每条染色体和每条染色体指定区域内的平均深度的总结。
2

下载安装
从官网直接下载编译好的文件,修改权限即可使用。

3

使用
准备

计算深度
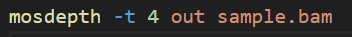
参数
● –t:设定自己需要的线程数。
● –c:设定待分析的染色体号。
● out:待输出的文件前缀。
● sample.bam:待输入分析的bam文件。
● –b sample.bed:待输入的指定区域文件,当分析整个基因组时,可去掉该参数。
● –n:不逐个输出碱基深度。如果跳过此输出,可以显著提升程序执行速度。建议优先使用量化或阈值的深度值进行测试。
● –f:用于CRAM文件的参考序列(fasta格式)。
●--d4:将碱基深度文件输出为d4格式。
4

结果
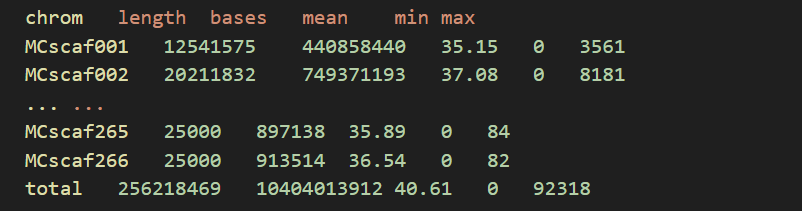
out.mosdepth.summary.txt
该文件详细记录了每条染色体及整个基因组的相关信息,包括以下内容:染色体名称、序列长度、映射的碱基数量、覆盖深度的平均值、覆盖深度的最小值以及最大值。这些数据能够全面反映基因组中不同区域的测序覆盖情况。以下是一个示例格式:
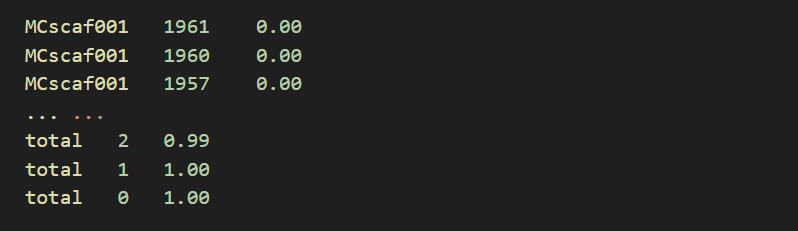
out.mosdepth.global.dist.txt
文件中包含累积分布信息,用于表示在不同覆盖率阈值下,被覆盖的总碱基比例。该文件包含三列数据,分别是:染色体名称或 total(表示全基因组统计)、覆盖深度阈值、以及达到或超过该覆盖深度的碱基比例。以下是一个示例:
还可以使用脚本 python scripts/plot-dist.py *global.dist.txt 绘制覆盖度分布图。此脚本会生成一个名为 dist.html 的输出文件,直观展示整个基因组的覆盖度分布情况。在运行该命令之前,请确保 scripts/plot-dist.py 脚本和覆盖度统计文件(以 *global.dist.txt 结尾)位于正确的目录中。生成的 dist.html 文件可在浏览器中打开,查看全基因组覆盖度分布的详细图示。
out.per-base.bed.gz
Mosdepth支持输出每个碱基的深度信息,即在基因组中对每个位置的覆盖深度进行逐一记录。这种输出方式提供了精确的深度分布数据,适用于需要详细覆盖度分析的场景,例如检测局部变异或基因热点区域。然而,由于逐碱基深度输出会生成大量数据,其处理和存储需求较高,因此在需要提高运行速度时,可以通过跳过此类输出(使用 -n 参数)来优化执行效率。
out.per-base.bed.gz.csi
该文件是一个索引文件,与压缩的逐碱基深度文件 out.per-base.bed.gz 对应,用于加速数据的随机访问和查询操作。

电话:021-61283010
网址:www.bio-chain.com
邮箱:marketing@bio-chain.com
地址:上海市徐汇区虹漕路421号65幢303室
上海柏辰生物科技有限公司 版权所有 严禁复制 网站备案号:沪ICP备09099502号-1 沪公网安备 31010402002899号 网站建设:铭心科技



