新闻中心
NEWS
生信篇 | Unicycler:混合测序时代的细菌基因组组装利器
Unicycler 是一款专为细菌基因组设计的混合组装工具,由 Ryan Wick 博士团队开发。它巧妙结合了二代测序中短读长(Illumina)的高准确性与三代测序中长读长(PacBio/Oxford Nanopore)的跨重复优势,特别适用于高重复、高杂合或复杂结构的微生物基因组组装。同时,它也是一款可以独立完成二代测序基因组组装、三代测序基因组组装以及二三代测序数据混合组装的优秀生信软件,下文将着重介绍二三代测序数据混合组装。
相较于传统组装工具(如SPAdes、IDBA-UD),Unicycler 在解决细菌基因组中重复序列和质粒环状结构时表现卓越,尤其适合单菌分离样本或宏基因组分箱后的精细化组装。
1

软件功能亮点
1、混合组装引擎
短读长纠错:利用Illumina数据校正长读长的测序错误,提升组装准确性。
长读长桥接:通过PacBio/Nanopore长读长跨越重复区域,连接短读长无法覆盖的断裂区域。
2、自动化流程整合
内置Bowtie2比对与Pilon纠错,支持从原始数据到最终环化基因组的全流程自动化。
3、环状结构识别
自动检测染色体和质粒的环状结构,生成完整的闭环序列(若数据支持)。
4、灵活输入支持
兼容Illumina双端测序、PacBio CLR/CCS及Nanopore数据,适应不同实验设计需求。
2

算法核心解析
Unicycler 的算法设计融合了De Bruijn图与字符串图(String Graph)的优势,分三阶段实现高效组装:
1、短读长纠错与初步组装
De Bruijn图构建:将短读长拆分为k-mer,构建图结构,通过寻找欧拉路径生成初始Contig。
错误剔除:基于k-mer频率和一致性过滤低覆盖分支,避免测序噪声干扰。
2、长读长进行的图优化
长读长比对与纠错:将长读长比对至初始Contig,校正其测序错误并填充缺口。
字符串图构建:基于长读长的重叠关系构建字符串图,解决重复区域的路径歧义。
3、路径选择与环化处理
启发式搜索最优路径:结合读长覆盖度和拓扑结构选择最可能的路径,生成线性或环状Scaffold。
自动环化检测:通过比对末端重叠识别环状结构,输出完整染色体/质粒序列。
3

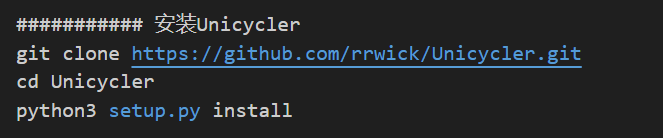
软件安装
Github官网上有提供相应源码,可直接下载安装,下面提供一种安装方案,可以安装最新版的Unicycler。
4

实战示例
案例背景:对一株耐药性的大肠杆菌进行基因组组装,数据源包含illumina双端测序(150bp)数据和Nanopore长读长(N50=15kb)数据。
1、运行命令

⭕ -1/-2:illumina 双端数据
⭕ - l:长读长数据
⭕ - o:输出目录
2、输出结果文件
⭕ assembly.fasta: 最终组装序列
⭕ assembly.gfa: 组装图文件,可用Bandage软件来可视化
⭕ unicycler.log:详细日志文件,可用于调试与性能评估
5

结果解读与优化策略
1、评估指标
Contig N50:若N50接近基因组预期大小(如大肠杆菌~4.6Mb),表明组装连贯性高。
环化比例:理想情况下,主染色体和质粒应标记为环状。
BUSCO完整性:使用细菌通用单拷贝基因集评估基因区域的完整性(目标>95%)。
2、常见问题与对策
碎片化Contig:增加长读长数据中的覆盖度或调整--min_kmer_coverage参数。
环化失败:检查长读长是否跨越重复区域,或手动使用Circlator等工具进行补环。
嵌合体污染:结合参考基因组比对或基于覆盖度差异筛选异常区域进行后续分析。
6

结语
Unicycler 凭借其混合算法与自动化设计,已成为微生物基因组研究的标杆工具。无论是临床病原体溯源还是环境微生物挖掘,它都能提供高完整度、低错误率的组装结果。下一期我们将带来Unicycler单独进行二代测序数据或者三代测序数据组装的详细介绍,敬请期待!

电话:021-61283010
网址:www.bio-chain.com
邮箱:marketing@bio-chain.com
地址:上海市徐汇区虹漕路421号65幢303室
上海柏辰生物科技有限公司 版权所有 严禁复制 网站备案号:沪ICP备09099502号-1 沪公网安备 31010402002899号 网站建设:铭心科技



