新闻中心
NEWS
生信篇 | 三代测序质控:NanoFilt vs Filtlong,谁更胜一筹?
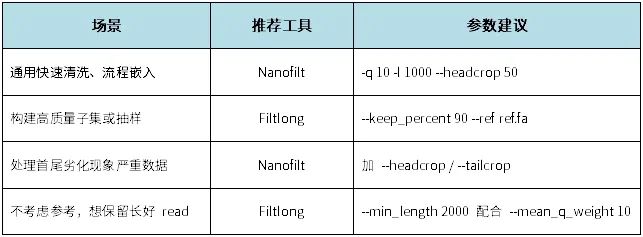
三代测序质控: NanoFilt vs Filtlong谁更胜一筹? ” 三代测序(尤其是 Nanopore)数据中普遍存在读长分布广、质量波动大等问题,质控作为下游比对和组装的前置步骤,其重要性不言而喻。本文聚焦两款高频使用的三代测序质控工具:NanoFilt 与 Filtlong,对比其原理、参数设置与实际表现,为你筛选最优方案。 1 工具简介 Nanofilt 使用Python编写,支持流式处理(stdin/stdout) 适用于快速过滤 Nanopore/PacBio 数据 支持设置平均质量、最小长度、首尾裁剪等参数 Filtlong 使用C++开发,效率更高 核心理念是打分排序 + 截留最优子集 可结合参考序列选择对参考更接近的 reads 2 参数设置与命令示例 Nanofilt 常用参数 -q:最低平均质量(Phred) -l:最小read长度 --headcrop:去除首端碱基(清理起始低质区域) --tailcrop:去除尾端碱基(去除末端漂移或污染) 🌟为什么要设置 --headcrop / --tailcrop? 因为三代测序因信号捕获机制,首尾碱基常存在质量劣化或接头残留,容易误导下游分析。合理裁剪首尾可有效提升 reads 整体质量,降低比对错误率。 Filtlong 常用参数 --min_length:最小保留长度 --keep_percent:保留打分靠前的 top n% --target_bases:保留指定总碱基量(适合抽样) --mean_q_weight:调整评分时对质量的权重 --ref:提供参考序列,根据比对得分筛选 reads(偏好真实信号) 3 两者比较 输入输出差异 Nanofilt:原生支持标准输入/输出(stdin/stdout),可无缝嵌入 | minimap2 | samtools 流式流程; Filtlong:支持从 stdin 读取数据,但输出默认写入文件,若需输出到 stdout 需显式使用 -,但流式处理兼容性相对较差,建议作为独立质控步骤使用。 各自优势 Nanofilt更侧重高效质量过滤,操作简洁且裁剪功能灵活多样; Filtlong更适合按目标碱基量筛选优质reads,支持基于参考的辅助优化; 若目标是精简且高质量子集,Filtlong 优势明显;反之若注重整体数据质量均衡,NanoFilt 更为便捷。 4 总结建议 5 技术提示 推荐使用 gunzip -c 解压 .gz 文件,避免 zcat 在某些系统上的兼容性问 NanoFilt 支持直接嵌入管道(| minimap2 | samtools)执行效率高 Filtlong 输出需显式控制格式,建议独立执行并输出到文件 可搭配 NanoStat 或 Filtlong --split 1 等方式评估质控效果 敬请关注,解锁更多生信实战干货!







电话:021-61283010
网址:www.bio-chain.com
邮箱:marketing@bio-chain.com
地址:上海市徐汇区虹漕路421号65幢303室
上海柏辰生物科技有限公司 版权所有 严禁复制 网站备案号:沪ICP备09099502号-1 沪公网安备 31010402002899号 网站建设:铭心科技



