新闻中心
NEWS
三代测序纠错全景解析:用算法还原真实的基因序列
01
三代测序的技术价值与纠错挑战
Values and Challenges
技术革新带来的颠覆性潜力
第三代测序(Third-Generation Sequencing, TGS)以超长读长(1 kb – 2 Mb)彻底重塑了基因组学研究的多个核心环节:
🔹跨越重复区域:解析端粒、着丝粒、大型结构变异等以往难以解析的复杂区域;
🔹全长转录本测定:直接获得完整的mRNA isoform结构,跳过拼接假设;
🔹实时病原检测:Oxford Nanopore可实现现场测序,广泛应用于疫情响应与临床快速诊断。
平台差异与错误机制
尽管TGS具备革命性的结构解析能力,但原始测序错误率远高于二代技术,纠错成为流程中不可或缺的一环。
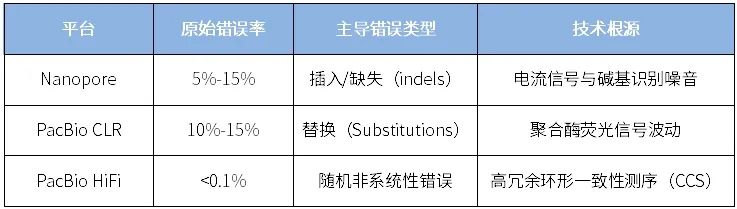
🔹Nanopore最新R10.4芯片 + Kit12试剂可将Indel错误率降低近40%(Nature, 2022);
🔹HiFi数据通常不需要传统纠错流程,可直接用于组装与变异检测,但仍可通过工具如DeepConsensus进一步提升质量。
02
纠错策略的三大主流路径
Error correction method
纠错方法大致可分为以下三类,每类针对不同的数据特征与分析目标。
自我纠错(Self-correction)
原理:利用长读段之间的冗余重叠区域,通过构建overlap consensus实现错误校正,适用于高深度测序场景(>30×)且无短读支持的项目。
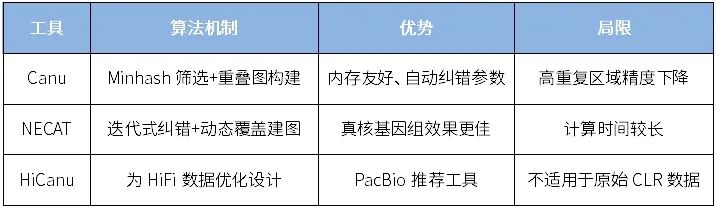
混合纠错(Hybrid correction)
原理:借助Illumina短读段的高准确度,从两个方向修正长读错误。
🔹k-mer图法(如LoRDEC):构建短读的de Bruijn图,对长读进行路径校正;
🔹比对共识法(如Proovread):直接将短读比对至长读并提取高置信区段。
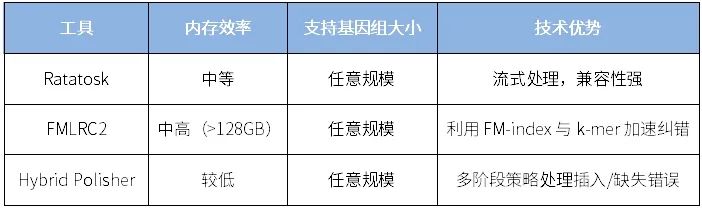
深度学习纠错(Deep learning-based)
原理:基于深度神经网络模型直接学习碱基误差特征,实现端到端的误差校正或碱基识别优化,已成为Nanopore和PacBio官方支持方向。
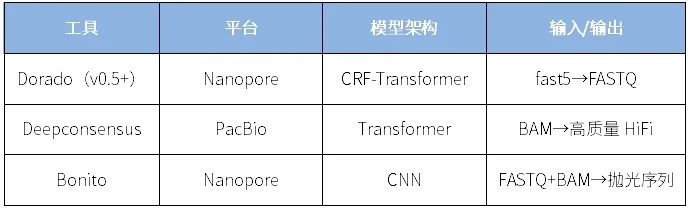
注意:Medaka不直接读取fast5原始信号。原始信号模型建议使用Bonito或Dorado。
03
纠错流程的分层设计
Process design
读段级纠错(Read-Level)
目标:提升原始读段质量
Nanopore标准流程:
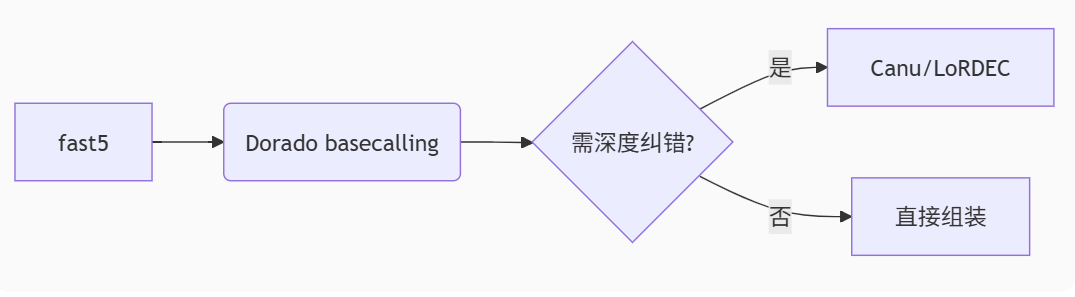
组装后抛光(Assembly Polishing)
目标:修正系统性错误
Nanopore 流程:

PacBio HiFi 流程:

04
纠错技术前景
Technical outlook
🔹算法融合:图神经网络(如2023年MetaFlye引入的模型)有望整合比对、图构建与深度学习;
🔹原始信号级纠错:直接对原始电流/脉冲信号建模,而非仅比对碱基;
🔹平台无关化:设计适配不同平台(PacBio HiFi vs ONT Q20+)的通用纠错模型。

扫描二维码关注我们
数字PCR平台|多重细胞因子检测平台|纳米孔测序平台

电话:021-61283010
网址:www.bio-chain.com
邮箱:marketing@bio-chain.com
地址:上海市徐汇区虹漕路421号65幢303室
上海柏辰生物科技有限公司 版权所有 严禁复制 网站备案号:沪ICP备09099502号-1 沪公网安备 31010402002899号 网站建设:铭心科技



